| 진행중인 프로젝트입니다. 작성중인 게시물입니다. |
밥을 잘 먹는 유전자를 가진 박테리아를 육성하는 시뮬레이터를 만들 것이다.
이 시뮬레이터는 유전 알고리즘을 기반으로 만들어진다.
실제로 박테리아는 유성생식을 하진 않지만, 유전물질을 교환하여 접합, 교차, 변이를 수행하기도 한다.
< 실제 생명공학의 유전 방법과는 다르다. 해당 알고리즘은 실제 현상을 모방하여 만들어졌을 뿐이다. >
lunaB/Genetic-Algorithm-Simulation
genetic algorithem simulator with HTML5 Canvas and Typescript - lunaB/Genetic-Algorithm-Simulation
github.com
요약
시각적으로나 알고리즘 적으로나 많이 개선해보려고 시도한 내용이다.

개선
개발 편에서 이미 유의미한 결과를 얻었기에 변형 편은 약간 번외 느낌으로 가려고 한다.
이것저것 변형시키고 개선시키는 과정을 거칠 것이다.
개선 TODO
- 시뮬레이팅 모드 개선
- 시뮬레이팅 시각화 개선
- 로그 변경
- 히스토리 추가

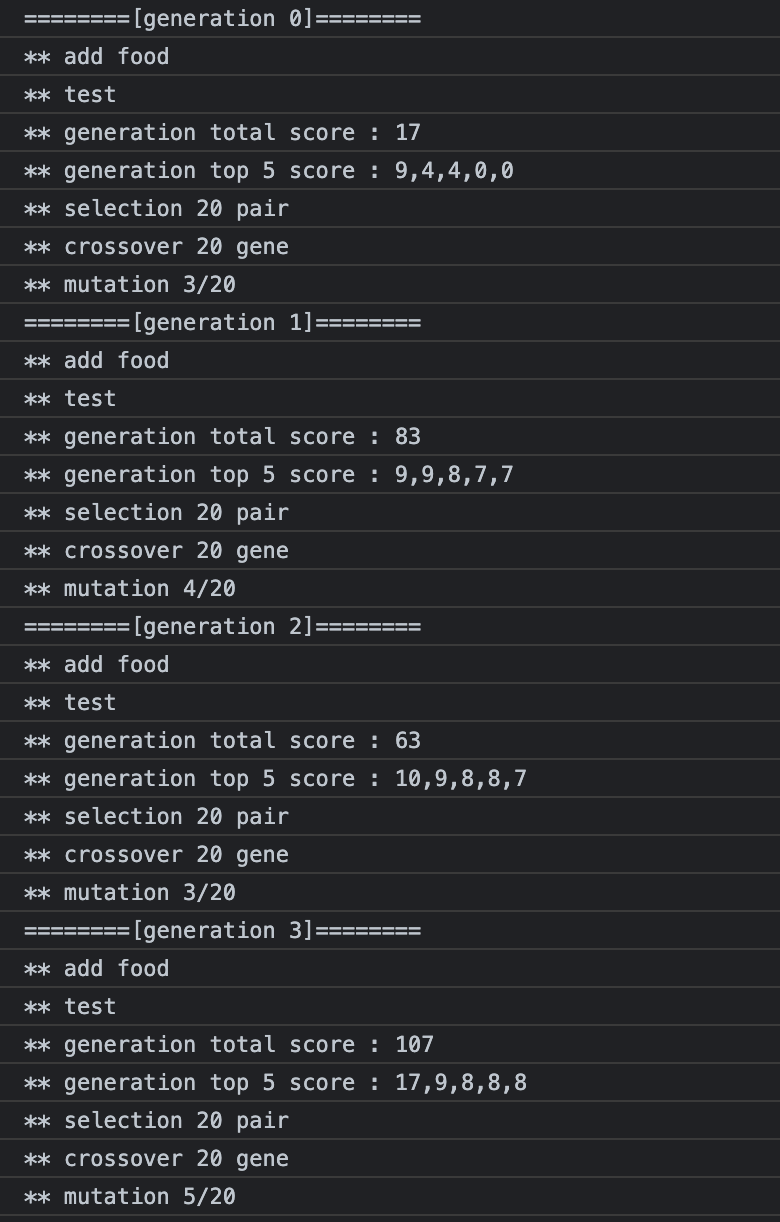
우선 시뮬레이터의 시각화 측면에서의 몇 가지 부분을 개선했다.
- 시뮬레이팅 모드
- 재생모드 : 모든 시뮬레이팅 과정을 정해놓은 프레임으로 감상할 수 있다. (30 프레임 정도가 적당한 것 같다.)
- 디버깅 모드 : 세대별로 약간씩 대기시간을 주고 마지막 화면만 업데이트한다. (위의 GIF가 디버깅 모드이다.)
- 연산 모드 : 시뮬레이터상에 업데이트하지 않고 연산과정만 수행한다. (조금 랙이 걸릴 수 있다)
빠른 연산이 필요할 때와, 느리게 지켜보는 게 필요할 때가 있다.
적절히 이용할 수 있도록 모드를 나누었다.
- 시뮬레이팅 시각화
- 시뮬레이팅 상황을 모니터링할 수 있는 차트
- 여러 라벨 데이터를 쉽게 추가할 수 있는 유틸
Chart.js를 이용했다. 모델 변경에 유연하게 작용하는 유틸을 추가했다.
처음에 d3.js로 개발했으나 코드가 너무 난잡해지는 느낌이 있어서 바꾸었다.
- 로그 변경
- document에 프린트되도록 했다.
기존에 사용하던 개발자 도구의 console.log()는 연산이 많은 작업에는 적합하지 않은 것 같다. 랙이 많이 걸려 불편하다.
당연한 말이지만 사실 웹에서 돌아가는 코드에 과한 연산을 부여하는 건 좋지 않다.
- 히스토리 추가
- 해당 시뮬레이팅 장면을 재현할 수 있는 히스토리 기능을 추가했다.
- 모델 변형과 검증 단계에서 더 수준 높은 유추를 할 수 있게 되었다.
강화 학습 모델을 만들 때도 히스토리 기능을 많이 사용하곤 한다. 우리의 적합 방식이랑 비슷한 부분이 많으니 분명 도움이 될 것이다.
히스토리 추가에 사용되는 메모리가 너무 높다 작게나마 레코딩 파일을 두어서 읽어올 수 있게 만드는 게 좋은 구조인 것 같다.
- 타입스크립트 구조적인 정비
- 자바스크립트로 만들다가 타입스크립트로 갈아탔다. 여러 가지 환경 조작에 유연하게 하기 위해서 타입에 혼동이 있으면 불편하기 때문이다.
- 타입스크립트 내부에서도 디자인 패턴을 고려하여 만들었다.
변형
문제점 분석
설계할 때의 목표는 화면 밖으로 안 나가는 박테리아 만들기였다.
로직상으로 그냥 못 나가게 막아버릴 수는 있지만,
환경과 보상을 잘 조작해주면 굳이 먹이가 없는 밖으로 나가지 않을 것이라고 생각했다.
하지만 가설일 뿐이고, 만들면서 생각이 든 문제점에 대하여 간단하게 분석해보려고 한다.
1. 어떤 위치에서 시작하느냐가 박테리아의 적합도 검증에 큰 영향을 미친다.
개발 편에서 언급했다시피 가장 유리한 방향으로 진동하며 이동하는 방향으로 진화했었다.
- 왼쪽에서 오른쪽으로 움직이도록 진화했다. 하지만 오른쪽에 스폰된다면?
2. 이동에 어떠한 외부 요인도 고려하지 않는다.
방향도 없다. 먹이 탐지도 없다.
적어도 박테리아가 높은 농도를 찾아가게 만들긴 해야 할 것 같다. - 이 부분은 다른 시뮬레이터를 만들 때 다른 기획으로 구현해볼 것이다.
3. 동족과 경쟁해야 한다.
동족이 먹이를 다 먹어버려도 문제다.
동족을 감지하는 로직은 넣지 않았다. - 이 부분은 다른 시뮬레이터를 만들 때 다른 기획으로 구현해볼 것이다.
유전자 수 증가, 매번 랜덤 유닛 5개 추가
20개의 유전자로는 표현될 수 있는 움직임이 너무 적다고 생각되었다.
20개면 최대로 많은 액션을 한다고 쳐도 (20-3)//2 = 8개다.
150개로 유전자 수를 크게 늘려보았다.
또한 매 세대에 랜덤 초기화 박테리아를 5개씩 추가했다.


작게나마 원형으로 회전하는 녀석이 생겼다. 생각보다 허무하게 목표가 이루어져서 다른 변형 로직도 넣어보려고 한다.
모든 박테리아 가운데서 스폰
가장 성능이 좋았던 유전자에서 계속 뿌리가 뻗어 나가기에
결국 모든 박테리아의 움직임은 하나의 패턴으로 수렴할 수밖에 없다.
빠르게 치고 나가 먹이를 최대한 많이 먹는 전략으로 수렴할 것이라고 생각했으나,
격하게 진동하여 다른 박테리아의 먹이를 뺏어 최고의 유전자가 되는 전략으로 움직였다.


경쟁 과정을 보길 기대하긴 힘들 것이라고 생각했는데 생각보다 쉽게 볼 수 있었다.
예상하지 못한 결과는 좋든 나쁘든 가슴 두근거리게 만든다.
탈출하면 감점! <번외>
맵 밖으로 탈출하지 않는 유전자를 만들기 위해 맵 밖으로 step마다 맵 밖에서 발견되면 evaluation을 감점 처리해봤다.
그 결과로 움직이지 않는 박테리아가 발견되었다. 미세조정이 필요해 보인다.
과식하면 사망 <번외>
과식하면 사망하게 만들면 적당히 먹을까?

얼추 평균적으로 5~9개로 많이 먹는 듯하다. 10개 이상 먹으면 과식으로 사망하게 해 보자.

적당히 먹으려고 최선을 다하는 박테리아의 모습을 볼 수 있다.